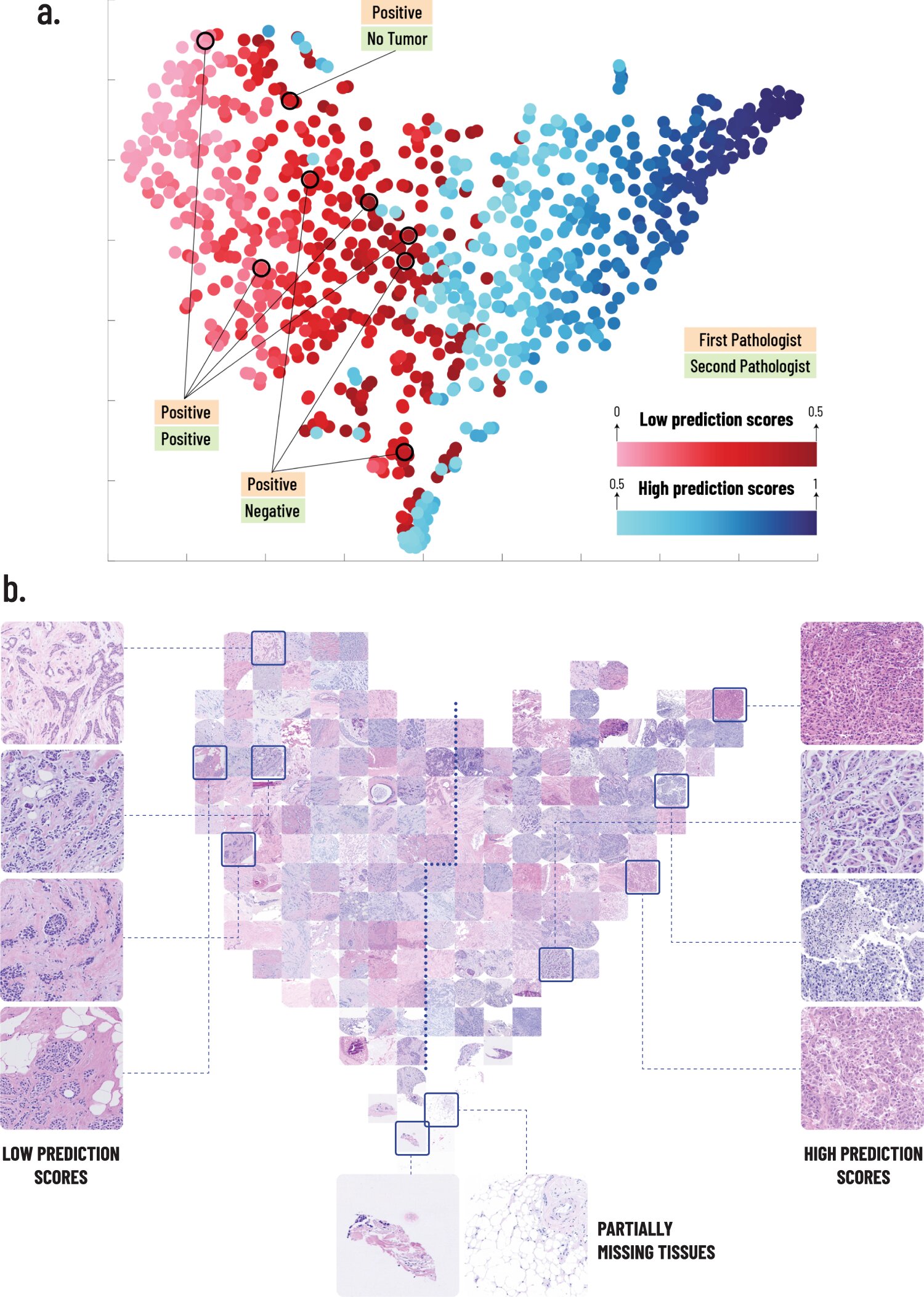
una. Una visualización 2D de los vectores de características de la imagen aplicando t-SNE. Cada punto representa un solo paciente en el conjunto de pruebas BCCA. La incorporación de t-SNE asigna pacientes con características de imagen similares a puntos cercanos y pacientes con características de imagen diferentes a puntos lejanos. Los puntos están coloreados por las puntuaciones de predicción de PD-L1 de sus pacientes correspondientes. Se marcan los 8 pacientes que fueron clasificados como positivos por el primer patólogo y bajo PS por el sistema y se anotan sus clasificaciones por ambos patólogos. b. Se presentan las imágenes TMA correspondientes a la incrustación t-SNE. Se muestran varios ejemplos de imágenes de puntuación de predicción alta y baja para demostrar las características observadas por los patólogos. En la parte inferior se muestran ejemplos de tejidos que faltan parcialmente. Crédito: Comunicaciones de la naturaleza (2022). DOI: 10.1038/s41467-022-34275-9
Una de cada nueve mujeres en el mundo desarrollado será diagnosticada con cáncer de mama en algún momento de su vida. La prevalencia del cáncer de mama está aumentando, un efecto causado en parte por el estilo de vida moderno y el aumento de la esperanza de vida. Afortunadamente, los tratamientos son cada vez más eficientes y personalizados. Sin embargo, lo que no aumenta, y de hecho disminuye, es el número de patólogos, o médicos cuya especialidad es examinar los tejidos del cuerpo para proporcionar el diagnóstico específico necesario para la medicina personalizada.
Por lo tanto, un equipo de investigadores del Technion-Israel Institute of Technology se ha propuesto convertir las computadoras en asistentes efectivos de los patólogos, simplificando y mejorando el trabajo de los médicos humanos. Su nuevo estudio fue publicado recientemente en Comunicaciones de la naturaleza.
La tarea específica que el Dr. Gil Shamai y Amir Livne del laboratorio del profesor Ron Kimmel de la Facultad de Ciencias de la Computación Henry and Marilyn Taub en Technion se propusieron lograr se encuentra dentro del ámbito de la inmunoterapia. La inmunoterapia ha ganado prominencia en los últimos años como un tratamiento eficaz, a veces incluso innovador, para varios tipos de cáncer. La base de esta forma de terapia es alentar al propio sistema inmunitario del cuerpo a atacar el tumor. Sin embargo, dicha terapia debe ser personalizada, ya que se debe administrar la medicación correcta a los pacientes que se beneficiarán de ella en función de las características específicas del tumor.
Múltiples mecanismos naturales evitan que nuestro sistema inmunológico ataque a nuestro propio cuerpo. Estos mecanismos a menudo son aprovechados por los tumores cancerosos para evadir el sistema inmunitario. Uno de esos mecanismos está relacionado con la proteína PD-L1: algunos tumores la muestran y actúa como una especie de contraseña al convencer erróneamente al sistema inmunitario de que el cáncer no debe ser atacado. La inmunoterapia específica para PD-L1 puede persuadir al sistema inmunitario para que ignore esta contraseña en particular, pero, por supuesto, solo sería eficaz cuando el tumor expresa PD-L1.
Es tarea de un patólogo determinar si el tumor de un paciente expresa PD-L1. Se utilizan marcadores químicos costosos para teñir una biopsia tomada del tumor a fin de obtener la respuesta. El proceso no es trivial, requiere mucho tiempo y, en ocasiones, es inconsistente. El Dr. Shamai y su equipo adoptaron un enfoque diferente. En los últimos años, se ha convertido en una práctica aprobada por la FDA escanear las biopsias para que puedan usarse para el análisis patológico digital. Amir Livne, el Dr. Shamai y el Prof. Kimmel decidieron ver si una red neuronal podría usar estos escaneos para hacer el diagnóstico sin requerir procesos adicionales. «Nos dijeron que no se podía hacer», dijo el equipo, «así que, por supuesto, teníamos que demostrar que estaban equivocados».
Las redes neuronales se entrenan de manera similar a como aprenden los niños: se les presentan múltiples ejemplos etiquetados. A un niño se le muestran muchos perros y varias otras cosas, ya partir de estos ejemplos se forma una idea de lo que es «perro». La red neuronal desarrollada por el equipo del Prof. Kimmel se presentó con imágenes de biopsias digitales de 3376 pacientes que fueron etiquetados como expresando o no expresando PD-L1. Después de la validación preliminar, se solicitó determinar si las imágenes de biopsia de ensayos clínicos adicionales de 275 pacientes eran positivas o negativas para PD-L1. Funcionó mejor de lo esperado: para el 70% de los pacientes, pudo determinar la respuesta con confianza y correctamente. Para el 30% restante de los pacientes, el programa no pudo encontrar los patrones visuales que le permitieran decidir de una forma u otra. Curiosamente, en los casos en que la inteligencia artificial (IA) no estuvo de acuerdo con la determinación del patólogo humano, una segunda prueba demostró que la IA tenía razón.
«Este es un logro trascendental», explicó el profesor Kimmel. «Las variaciones que encontró la computadora no son distinguibles para el ojo humano. Las células se organizan de manera diferente si presentan PD-L1 o no, pero las diferencias son tan pequeñas que incluso un patólogo capacitado no puede identificarlas con confianza. Ahora nuestro la red neuronal puede».
Este logro es el trabajo de un equipo compuesto por el Dr. Gil Shamai y el estudiante de posgrado Amir Livne, quien desarrolló la tecnología y diseñó los experimentos, el Dr. António Polonia del Instituto de Patología Molecular e Inmunología de la Universidad de Oporto, Portugal, Profesor Edmond Sabo y la Dra. Alexandra Cretu del Centro Médico Carmel en Haifa, Israel, que son patólogos expertos que realizaron la investigación, y con el apoyo del profesor Gil Bar-Sela, jefe de la división de oncología y hematología del Centro Médico Haemek en Afula, Israel .
«Es una oportunidad increíble para unir la inteligencia artificial y la medicina», dijo el Dr. Shamai. «Me encantan las matemáticas, me encanta desarrollar algoritmos. Ser capaz de usar mis habilidades para ayudar a las personas, para hacer avanzar la medicina, es más de lo que esperaba cuando comencé como estudiante de informática». Ahora dirige un equipo de 15 investigadores, que están llevando este proyecto al siguiente nivel.
«Esperamos que la IA se convierta en una poderosa herramienta en manos de los médicos», compartió el Prof. Kimmel. «La IA puede ayudar a hacer o verificar un diagnóstico, puede ayudar a adaptar el tratamiento al paciente individual, puede ofrecer un pronóstico. No creo que pueda, o deba, reemplazar al médico humano. Pero puede hacer algunos elementos de el trabajo de los médicos sea más simple, más rápido y más preciso».
Gil Shamai et al, El análisis de imágenes basado en el aprendizaje profundo predice el estado de PD-L1 a partir de imágenes histopatológicas teñidas con H&E en el cáncer de mama, Comunicaciones de la naturaleza (2022). DOI: 10.1038/s41467-022-34275-9
Citación: Con la disminución del número de patólogos, el equipo desarrolla IA para usarla como herramienta de diagnóstico (1 de diciembre de 2022) consultado el 1 de diciembre de 2022 de https://medicalxpress.com/news/2022-12-pathologists-declining-team-ai -diagnóstico.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.