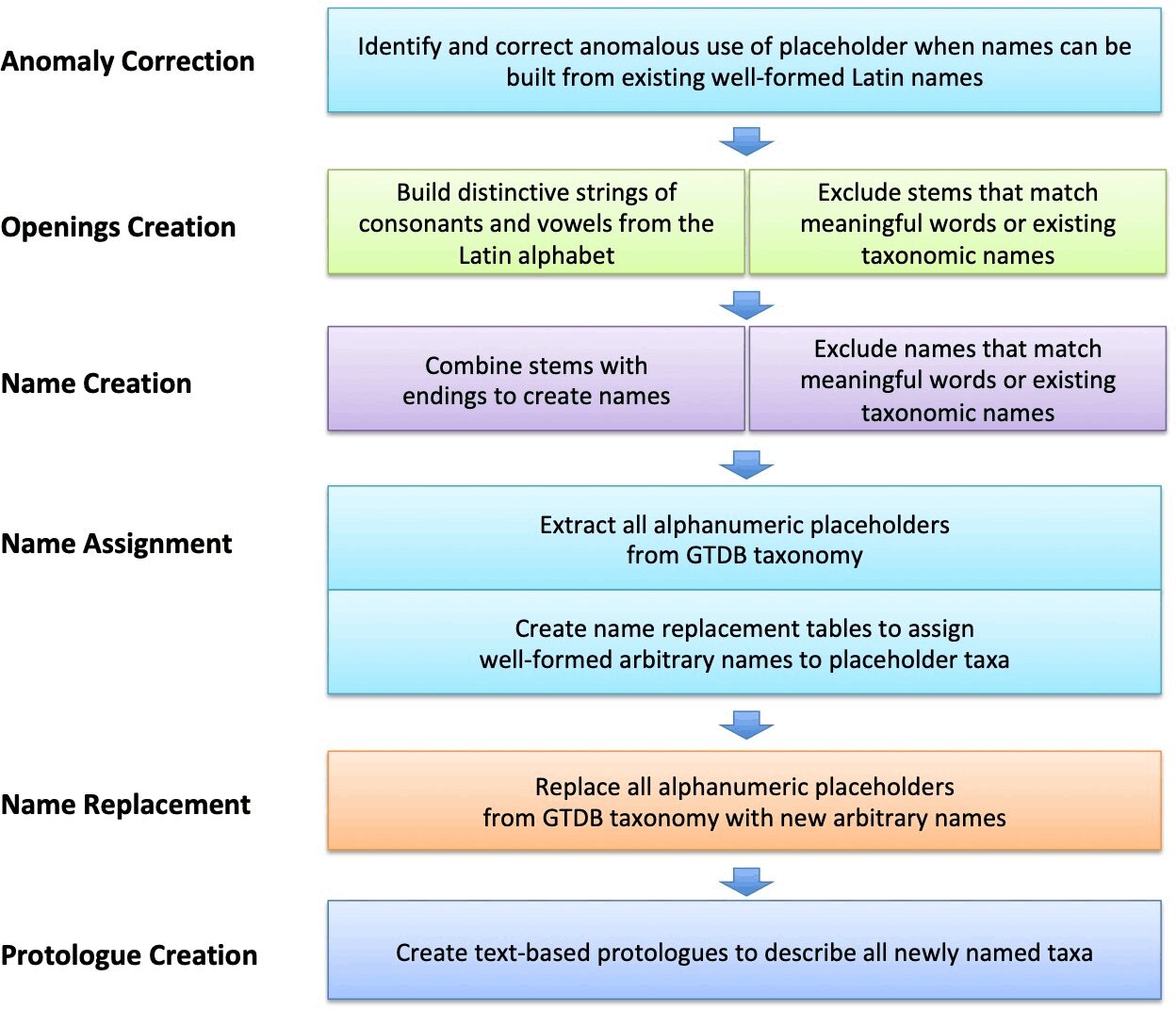
Flujo de trabajo esquemático para la creación y asignación de nombres latinos arbitrarios. Las cadenas construidas a partir de letras latinas se seleccionaron y curaron para excluir componentes no deseados y palabras significativas. Estos se combinaron con sufijos o palabras latinas para crear nombres, que luego se usaron para nombrar taxones de marcadores de posición en archivos GTDB y crear protólogos basados en texto. Crédito: Revista Internacional de Microbiología Sistemática y Evolutiva (2022). DOI: 10.1099/ijsem.0.005482
En un artículo publicado recientemente en el Revista Internacional de Microbiología Sistemática y Evolutiva, investigadores del Reino Unido y Austria han nombrado más de 65 000 tipos diferentes de microbios. El estudio, dirigido por el profesor Mark Pallen del Instituto Quadram en Norwich, se basa en una larga tradición de crear nombres latinos bien formados pero arbitrarios para nuevas especies, pero aplica este enfoque a una escala sin precedentes en la historia de la taxonomía.
El Prof. Pallen argumenta que la microbiología es víctima de su propio éxito, con decenas de miles de nuevas especies descubiertas en los últimos años, aunque la mayoría permanece sin nombre. En el pasado, las bacterias recibieron nombres descriptivos o recibieron nombres de personas o lugares. Este enfoque ofrece actualmente alrededor de mil nuevos nombres de especies por año. Sin embargo, con una acumulación de más de 50 000 especies bien clasificadas pero sin nombre, a este ritmo de progreso, se necesitaría al menos medio siglo para nombrar todas estas bacterias sin nombre, momento en el que los científicos se enfrentarían al problema de nombrar millones de especies más. descubierto mientras tanto.
Pallen y sus colegas han adoptado un enfoque eficiente, de alto rendimiento y de big data, utilizando un programa de computadora para generar decenas de miles de nombres distintivos pero fáciles de usar que no se parecen a ninguna palabra existente. Para cumplir con el requisito de que los nombres de las bacterias deben estar en latín, el equipo combinó cadenas arbitrarias de letras del alfabeto latino con sufijos femeninos gramaticalmente bien formados. El resultado es un conjunto de nombres que recuerdan la familiaridad y la seriedad del latín, aunque carecen de un pedigrí significativo. Los ejemplos incluyen Dupisella tifacia para una bacteria del intestino de las ovejas, Hopelia gocarosa para una bacteria que vive en aguas subterráneas suecas o Saxicetta apufaria de un lago salado ruso.
Aunque esto pueda parecer radical, de hecho, la formación de nombres de manera arbitraria tiene una larga tradición, que se remonta al primer código de nomenclatura taxonómica de 1869 e incluso al padre de la taxonomía, el naturalista sueco Linneo. Lo que es diferente aquí es el asombroso sentido de la escala, con un catálogo de nombres que se extiende por más de diez mil páginas en lo que representa el nombre más grande de especies en una sola publicación.
Debido a que se han aplicado a especies descubiertas por secuenciación de ADN pero que aún no se cultivan en el laboratorio, por ahora los nuevos nombres siguen siendo provisionales y no permanentes. Sin embargo, dado que el código de nomenclatura insiste en que los bacteriólogos buscan la estabilidad en los nombres y no existen enfoques competitivos de denominación a escala, parece probable que la gran mayoría de los nuevos nombres se utilicen durante años o incluso siglos por venir.
Para finalizar, Pallen dice: «Fui miembro del grupo de trabajo que nos proporcionó letras griegas para las variantes de COVID, que fueron adoptadas rápidamente por la comunidad científica. Espero que los nombres propuestos aquí también se adopten rápidamente y se utilicen ampliamente. Esto es solo el primer paso. La era del descubrimiento microbiano está lejos de terminar, pero será fácil crear nombres futuros en masa utilizando los principios que hemos establecido aquí».
Un sistema automatizado para generar un millón de nuevos nombres para bacterias
Mark J. Pallen et al, Nombrar a los sin nombre: más de 65 000 nombres de Candidatus para arqueas y bacterias sin nombre en la base de datos de taxonomía del genoma, Revista Internacional de Microbiología Sistemática y Evolutiva (2022). DOI: 10.1099/ijsem.0.005482
Proporcionado por el Instituto Quadram
Citación: Nombrar especies de bacterias sin nombre en la era de los grandes datos (21 de septiembre de 2022) recuperado el 21 de septiembre de 2022 de https://phys.org/news/2022-09-unnamed-species-bacteria-age-big.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.